在日常的建模过程中常常需要特征进行筛选,选择与模型相关度最高的特征,避免过拟合。通常使用的最多的方法是决策树中的feature_importance。
scikit-learn决策树
scikit-learn决策树类中的feature_importances_属性返回的是特征的重要性,feature_importances_越高代表特征越重要。feature_importances_属性,返回的重要性是按照决策树中被用来分割后带来的增益(gain)总和进行返回。
示例代码:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import GradientBoostingClassifier
from sklearn.ensemble import ExtraTreesClassifier
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, test_size=0.3)
# dtc = DecisionTreeClassifier()
# dtc.fit(X_train, y_train)
# importances = dtc.feature_importances_
rfc = RandomForestClassifier()
rfc.fit(X_train, y_train)
importances = rfc.feature_importances_
indices = np.argsort(importances)[::-1]
for f in range(X_train.shape[1]):
print("%2d) %-*s %f" % (f+1, 30, wine['feature_names'][f], importances[indices[f]]))
plt.title('Feature Importance')
plt.bar(range(X_train.shape[1]), importances[indices], color='lightblue', align='center')
plt.xticks(range(X_train.shape[1]), wine['feature_names'], rotation=90)
plt.xlim([-1, X_train.shape[1]])
plt.tight_layout()
plt.show()
输出:
1) alcohol 0.171172 2) malic_acid 0.157900 3) ash 0.147470 4) alcalinity_of_ash 0.130593 5) magnesium 0.103220 6) total_phenols 0.090347 7) flavanoids 0.049028 8) nonflavanoid_phenols 0.043786 9) proanthocyanins 0.026869 10) color_intensity 0.026426 11) hue 0.025300 12) od280/od315_of_diluted_wines 0.015860 13) proline 0.012030
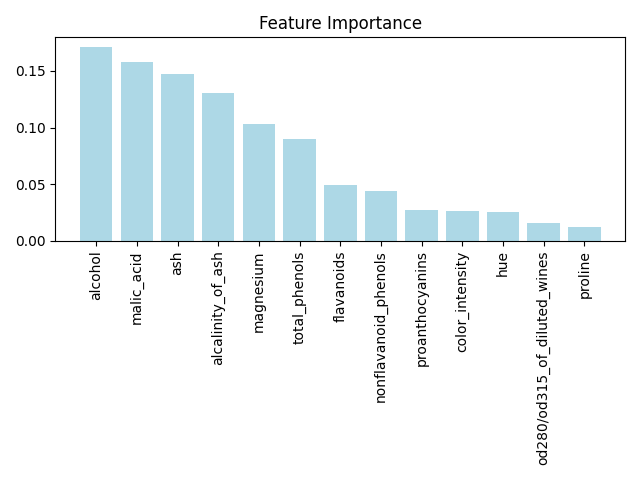
在skleran中不管是分类还是回归,主要是决策树类型的算法都可以使用上述方法。
XGBoost
XGBoost同样是基于决策树的算法。XGboost同样的存在feature_importances_属性。与scikit-learn中决策树不同的是feature_importances_输出的重要性与模型超参数设置的importance_type相关。importance_type可选值:
- weight:该特征被选为分裂特征的次数。
- gain:该特征的带来平均增益(有多棵树)。在tree中用到时的gain之和/在tree中用到的次数计数。gain=total_gain/weight
- cover:该特征对每棵树的覆盖率。
- total_gain:在所有树中,某特征在每次分裂节点时带来的总增益
- total_cover:在所有树中,某特征在每次分裂节点时处理(覆盖)的所有样例的数量。
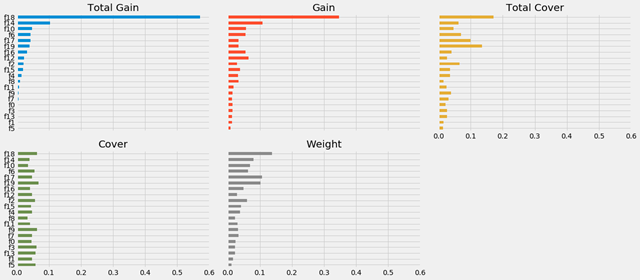
另外XGBoost提供了一个内置的plot_importance()方法可按重要性绘制特征。
import matplotlib.pyplot as plt
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
from xgboost import plot_importance
from xgboost import XGBClassifier
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, test_size=0.3)
cls = XGBClassifier(importance_type='gain')
cls.fit(X_train, y_train)
cls.get_booster().feature_names = wine['feature_names']
importances = zip(wine['feature_names'], cls.feature_importances_)
for f in importances:
print(f)
plt.barh(wine['feature_names'], cls.feature_importances_)
plt.show()
plot_importance(cls, importance_type='gain')
plt.show()
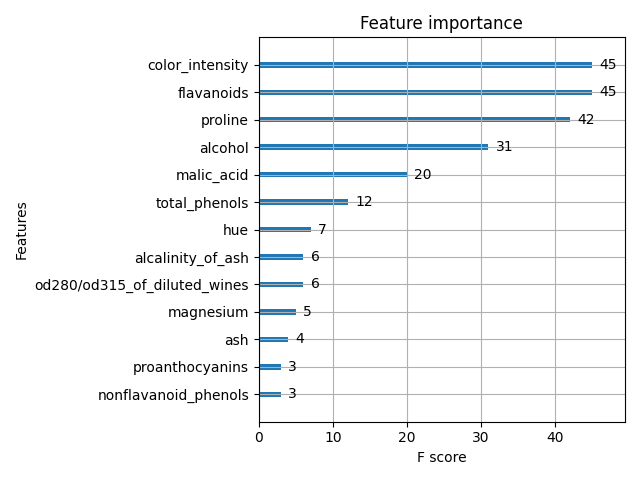
LightGBM
LightGBM同样是决策树算法。LightGBM同样提供feature_importances_属性,使用 feature_importances_ 之前需要设置模型超参数 importance_type。与 xgboost 不同的是这里只有 2 个选项:
- split 就是特征在所有决策树中被用来分割的总次数。(默认)
- gain 就是特征在所有决策树种被用来分割后带来的增益(gain)总和
另外 LightGBM 提供feature_importance() 方法,效果同 feature_importances_。lightgbm 也提供plot_importance() 方法直接绘图。
LightGBM 可以计算两种不同类型的特征重要性:基于分裂(Split-based)和基于增益(Gain-based)。
基于分裂的特征重要性是根据特征在决策树中被使用的次数来计算的。具体来说,它测量了当一个特征在决策树的分裂中被使用时,该特征能够降低多少决策树的不纯度(例如,Gini 系数或熵)。这种方法更快速,但可能会出现过拟合的情况,特别是在树的深度很浅时。
假设一个极端的场景,某一个二值特征,性别,假设我们有 100 个样本,二分类问题,好坏比例 10:1 吧,即 90 个 0 和 10 个 1,假设我们使用性别进行分裂(显然性别这个特征在 tree 中只能分裂一次),并且假设性别这个特征的区分度很高,一次切分,左枝一共 50 个用户,全是 0,右枝 50 个样本,40 个 0 和 10 个 1,假设后续的特征都持续分裂直到每个 1 都被分裂到一个单独的叶子节点,即分裂次数很多,除了性别之外,其它的特征可能会被分裂多次,则根据 split 来计算,我们会发现性别的 split 的 feature importances 非常低,然而实际上这个特征是强特,它的 gain 很高;因此,一般情况下建议使用 gain 而不是 split,split 的方式会出现上述的问题。
基于增益的特征重要性是根据特征对目标变量的预测能力来计算的。具体来说,它测量了当一个特征在决策树的分裂中被使用时,该特征能够提高多少模型的性能(例如,平均准确率或平均精确度)。这种方法更准确,但需要更多的计算资源。
因此,选择使用哪种特征重要性取决于你对模型性能和计算资源之间的权衡。如果你想快速计算特征重要性并不太关心模型的精确度,则可以使用基于分裂的特征重要性。如果你想获得更准确的特征重要性并且有足够的计算资源,则可以使用基于增益的特征重要性。
代码示例:
import matplotlib.pyplot as plt
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
import lightgbm as lgb
import seaborn as sns
import pandas as pd
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, test_size=0.3)
cls = lgb.LGBMClassifier(importance_type='gain')
cls.fit(X_train, y_train)
#importances = zip(wine['feature_names'], cls.feature_importances_)
#for f in importances:
#print(f)
importances = zip(wine['feature_names'], cls.booster_.feature_importance(importance_type='gain'))
for f in importances:
print(f)
feature_imp = pd.DataFrame(sorted(zip(cls.feature_importances_, wine['feature_names'])), columns=['Value','Feature'])
plt.figure(figsize=(20,10))
sns.barplot(x="Value", y="Feature", data=feature_imp.sort_values(by="Value", ascending=False))
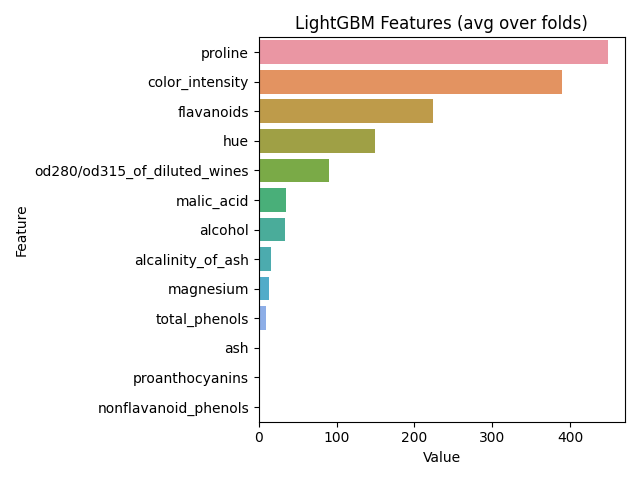
plt.title('LightGBM Features (avg over folds)')
plt.tight_layout()
plt.show()
lgb.plot_importance(cls.booster_, importance_type='gain')
plt.show()